- 線形混合効果モデル(こちら)
- パッケージの説明PDF
- メタアナリシスのときに、複数のスタディに等しいリスクを想定するのがFixed effectモデルで、異なるリスクを想定するのがrandom effectモデルですが、線形回帰のときに、ベースラインや、回帰直線の傾きについて、群ごとに同じで考えれば、Fixed、群ごとに違う値を考えればRandom。線形混合効果モデルでも、"Fixed effect","Random effect"がそういう文脈で使われています。
- 前置き:lmer()が挙動不審とのコメントあり。要注意(こちら)
- ランダム・エフェクトを切片にのみでよければ、glmmML() in library(glmmML)でも
- さて、4時刻にわたって、測定されたデータがあるものとする
- 横軸は時刻0,1,6,60が等間隔
- 縦軸は観測値
- 全体の傾向としては、明らかに右肩上がり
- 3色は、後述する3ジェノタイプで塗り分け
- 初期値が高めだけれど、傾きが小さいか、初期値が低めだけれど、傾きが大きいか、で3ジェノタイプには明らかな違いがある
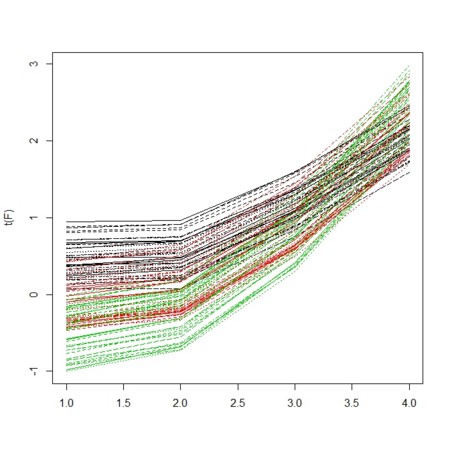
- その4時刻には、1次線形なトレンドがあることが間違いないものとする
- ここで、この1次線形なトレンドが、標本の属性(たとえば、2値型遺伝子多型のディプロタイプ)に関して(相加モデルで)影響されているかどうかを調べたいとする
- 標本の属性は、観測値のベースラインに影響する(経時的測定だが、その時刻によらず、常に同じ影響を与えている)かもしれない
- 標本の属性は、観測値のベースラインに影響を与えるとともに、経時的変化の傾きにも影響を与えているかもしれない
- こんなことを調べたい
- 次のようにする
library(lme4)
Ns<-100
nT<-4
T<-c(0,1,6,60)
I<-1:Ns
I<-paste("",I)
G<-sample(0:2,Ns,replace=TRUE)
A<-sample(30:80,Ns,replace=TRUE)
B<-sample(0:1,Ns,replace=TRUE)
S<-cbind(I,G,A,B)
F<-matrix(0,Ns,Nt)
F[,1]<-runif(Ns)-0.5*G
F[,2]<-F[,1]+0.1*G+runif(Ns)*0.1
F[,3]<-F[,2]+0.2*(G+3)+runif(Ns)*0.1
F[,4]<-F[,3]+0.4*(G+2)+runif(Ns)*0.1
matplot(t(F),type="l",col=(G+1))
Fv<-c(F)
-
- サンプル属性データの行数を観測項目のそれにそろえる
- 時刻も加える
Sv<-as.data.frame(S)
for(i in 2:Nt){
Sv<-rbind(Sv,S)
}
T<-c(matrix(rep(t,Ns),byrow=TRUE,ncol=Nt))
Sv<-cbind(Sv,T)
m1out<-lmer(Fv~Sv$T+Sv$A+Sv$B+(1|Sv$I))
m2out<-lmer(Fv~Sv$T+Sv$A+Sv$B+(1|Sv$G)+(1|Sv$I))
m3out<-lmer(Fv~Sv$T+Sv$A+Sv$B+(Sv$T|Sv$G)+(1|Sv$I))
m2out
fixef(m2out)
ranef(m2out)
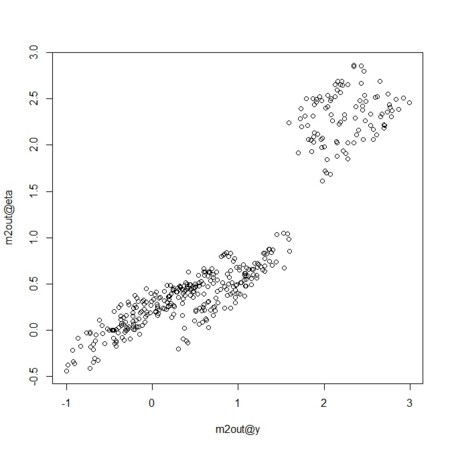
plot(m2out@y,m2out@eta)
-
- 結果をanovaで検定する
- 変数を増やしたモデルを増やす前のモデルに対して比較している
anova(m1out,m2out)
anova(m2out,m3out)
> anova(m1out,m2out)
Data:
Models:
m1out: Fv ~ Sv$T + Sv$A + Sv$B + (1 | Sv$I)
m2out: Fv ~ Sv$T + Sv$A + Sv$B + (1 | Sv$G) + (1 | Sv$I)
Df AIC BIC logLik Chisq Chi Df Pr(>Chisq)
m1out 51 584.31 787.88 -241.16
m2out 52 569.42 776.98 -232.71 16.887 1 3.968e-05 ***
---
Signif. codes: 0 ‘***’ 0.001 ‘**’ 0.01 ‘*’ 0.05 ‘.’ 0.1 ‘ ’ 1
> anova(m2out,m3out)
Data:
Models:
m2out: Fv ~ Sv$T + Sv$A + Sv$B + (1 | Sv$G) + (1 | Sv$I)
m3out: Fv ~ Sv$T + Sv$A + Sv$B + (Sv$T | Sv$G) + (1 | Sv$I)
Df AIC BIC logLik Chisq Chi Df Pr(>Chisq)
m2out 52 569.42 776.98 -232.71
m3out 54 462.63 678.17 -177.31 110.8 2 < 2.2e-16 ***
---
Signif. codes: 0 ‘***’ 0.001 ‘**’ 0.01 ‘*’ 0.05 ‘.’ 0.1 ‘ ’ 1
-
- 個人ごとの寄与を完全に個人をばらばらにしたが、GWAS-PCAの情報などを使って、主要軸を取りこんでもよい
Na<-3
P<-matrix(rnorm(Na*Ns),ncol=Na)
Pv<-P
for(i in 2:Nt){
Pv<-rbind(Pv,P)
}
m1out.P<-lmer(Fv~Sv$T+Sv$A+Sv$B+(1|Pv[,1])+(1|Pv[,2])+(1|Pv[,3]))
m2out.P<-lmer(Fv~Sv$T+Sv$A+Sv$B+(1|Sv$G)+(1|Pv[,1])+(1|Pv[,2])+(1|Pv[,3]))
m3out.P<-lmer(Fv~Sv$T+Sv$A+Sv$B+(Sv$T|Sv$G)+(1|Pv[,1])+(1|Pv[,2])+(1|Pv[,3]))
anova(m1out.P,m2out.P)
anova(m2out.P,m3out.P)
-
- すべてのサンプルに全時刻の情報がなくてもよい
- サンプルごとに、時系列の切片と傾きを引く処理だから、観測時刻がばらばらでもよい
selected.time<-sample(1:length(Fv),length(Fv)/2)
Fv.s<-Fv[selected.time]
Sv.s<-Sv[selected.time,]
Sv.s$T<-Sv.s$T*rnorm(length(selected.time),1,0.2)
plot(Sv$T[selected.time],Sv.s$T)
m1out.s<-lmer(Fv.s~Sv.s$T+Sv.s$A+Sv.s$B+(1|Sv.s$I))
m2out.s<-lmer(Fv.s~Sv.s$T+Sv.s$A+Sv.s$B+(1|Sv.s$G)+(1|Sv.s$I))
m3out.s<-lmer(Fv.s~Sv.s$T+Sv.s$A+Sv.s$B+(Sv.s$T|Sv.s$G)+(1|Sv.s$I))
anova(m1out.s,m2out.s)
anova(m2out.s,m3out.s)
glmer(Fv~Sv$T+Sv$A+Sv$B+(1|Sv$G)+(1|Pv[,1])+(1|Pv[,2])+(1|Pv[,3]),family=gaussian)

