- 昨日の記事で、BRACAPROとかその姉妹版の話を書いた
- その背景には家系データを扱うという、「マイ・ブーム」がある(こちら)
- 以前、法医学関係でベイジアンネットワークについて書いたので、復習を兼ねて、少数ローカスがリスクを運び、それによって発病年齢が影響を受ける場合の家系データのベイジアンネットワークを作ってみたい(できるかどうかわからないけれど)
- まずは、その地ならし
- 複数ローカスが作る複合ジェノタイプを両親に持たせて、そのペアから子のジェノタイプの生起確率の表を任意のローカス数でさらさらと作りたい
- 伝達関係は0,1/2,1のメンデルがローカスの数だけ重層化
oya2.1 <- array(0,rep(3,3))
oya2.1[1,1,1] <- oya2.1[2,1,3] <- oya2.1[2,3,1] <- oya2.1[3,3,3] <- 1
oya2.1[1,1,2] <- oya2.1[1,2,1] <- oya2.1[2,1,2] <- oya2.1[2,2,1] <- oya2.1[2,2,2] <- oya2.1[2,2,3] <- oya2.1[2,3,2] <- oya2.1[3,2,3] <- oya2.1[3,3,2] <- 1/2
oya2.1[1,2,2] <- oya2.1[3,2,2] <- 1/4
oya2 <- array(0,rep(n.g,3))
for(i in 1:n.g){
father <- unlist(g[i,])
for(j in 1:n.g){
mother <- unlist(g[j,])
tmp.list <- list()
tmp.list[[1]] <- oya2.1[,father[1]+1,mother[1]+1]
if(n.l>1){
for(k in 1:n.l){
tmp.list[[k]] <- oya2.1[,father[k]+1,mother[k]+1]
}
}
tmp <- tmp.list[[1]]
if(n.l>1){
for(k in 2:n.l){
tmp <- outer(tmp,tmp.list[[k]],"*")
}
}
oya2[i,j,] <- c((tmp))
}
}
oya2.tandem <- matrix(0,0,n.g)
for(i in 1:n.g){
oya2.tandem <- rbind(oya2.tandem,oya2[i,,])
}
oya2.tandem <- c(t(oya2.tandem))
g <- 0:2
one.locus <- array(0,rep(3,3))
for(i in 1:3){
for(j in 1:3){
if(i==1 & j==1){
one.locus[,i,j] <- c(1,0,0)
}else if(i==1 & j==2){
one.locus[,i,j] <- c(1/2,1/2,0)
}else if(i==1 & j==3){
one.locus[,i,j] <- c(0,1,0)
}else if(i==2 & j==1){
one.locus[,i,j] <- c(1/2,1/2,0)
}else if(i==2 & j==2){
one.locus[,i,j] <- c(1/4,1/2,1/4)
}else if(i==2 & j==3){
one.locus[,i,j] <- c(0,1/2,1/2)
}else if(i==3 & j==1){
one.locus[,i,j] <- c(0,1,0)
}else if(i==3 & j==2){
one.locus[,i,j] <- c(0,1/2,1/2)
}else{
one.locus[,i,j] <- c(0,0,1)
}
}
}
n.l <- 2
g.complex <- expand.grid(rep(list(g),n.l))
n.g <- length(g.complex[,1])
oyako <- array(0,rep(n.g,3))
g.comp.pair <- expand.grid(1:n.g,1:n.g)
values <- c()
for(i in 1:length(g.comp.pair[,1])){
oya1.compg <- g.comp.pair[i,1]
oya2.compg <- g.comp.pair[i,2]
oya1.g <- unlist(g.complex[oya1.compg,])
oya2.g <- unlist(g.complex[oya2.compg,])
per.locus <- list()
for(k in 1:length(oya1.g)){
per.locus[[k]] <- one.locus[,oya1.g[k]+1,oya2.g[k]+1]
}
tmp <- as.matrix(expand.grid(per.locus),ncol=length(oya1.g))
values <- c(values,apply(tmp,1,prod))
}
for(i in 1:n.g){
oya1 <- g.complex[i,]
for(j in 1:n.g){
oya2 <- g.complex[j,]
per.locus <- list()
for(k in 1:length(oya1)){
per.locus[[k]] <- one.locus[,oya1+1,oya2+1]
}
}
}
- このネットワークには、すべての個人のジェノタイプのフェノタイプに対応するノードがある
- さらに、一般集団個人として2人のジェノタイプノードがある。これは家系図において親がいない個人の仮想的親のジェノタイプノードである
- 子のジェノタイプは親のジェノタイプに依存する
- 個人のフェノタイプは個人のジェノタイプに依存する
- 個人のフェノタイプは個人の年齢と個人のジェノタイプによるので、個人のフェノタイプ・ジェノタイプ関係のテーブルは年齢ごとに異なるものとする
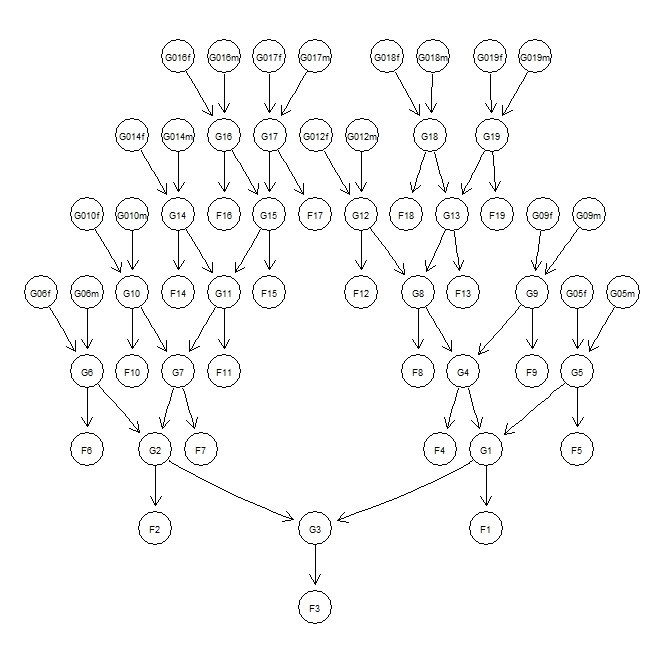
my.pedigree <- function(n.init=3,n.iter=20,mean.kid=2){
trio <- matrix(0,n.init,5)
m <- length(trio[1,])
trio[1:n.init,1] <- 1:n.init
trio[1:n.init,4] <- sample(c(-1,1),n.init,replace=TRUE)
trio[,5] <- 0
cnt <- n.init
tmp.f <- function(m,k){
(m/(1-exp(-m))-k)^2
}
xmin <- optimize(tmp.f, c(0, mean.kid), tol = 0.0001, k = mean.kid)
mean.kid. <- xmin[[1]]
for(i in 1:n.iter){
s <- sample(trio[,1],1)
if(length(which(trio[,2:3]==s))==0){
trio <- rbind(trio,c(cnt+1,rep(0,m-1)))
dp <- dpois(1:100,mean.kid.)
dp <- dp/sum(dp)
num.kids <- sample(1:100,1,prob=dp)
tmp <- matrix(0,num.kids,m)
tmp[,1] <- cnt+1+(1:num.kids)
if(trio[s,4]==1){
tmp[,2] <- s
tmp[,3] <- cnt+1
}else{
tmp[,2] <- cnt+1
tmp[,3] <- s
}
tmp[,4] <- sample(c(-1,1),num.kids,replace=TRUE)
tmp[,5] <- trio[s,5] + 1
trio <- rbind(trio,tmp)
trio[cnt+1,4] <- -trio[s,4]
trio[cnt+1,5] <- trio[s,5]
cnt <- cnt+1+num.kids
}else if(trio[s,2] == 0 & trio[s,3] == 0){
trio <- rbind(trio,c(cnt+1,rep(0,m-1)))
trio <- rbind(trio,c(cnt+2,rep(0,m-1)))
trio[cnt+1,4] <- 1
trio[cnt+2,4] <- -1
trio[c(cnt+1,cnt+2),5] <- trio[s,5]-1
trio[s,2] <- cnt+1
trio[s,3] <- cnt+2
cnt <- cnt+2
}
}
trio
}
make.genotype <- function(trio,prob){
ret <- rep(0,length(trio[,1]))
ord <- order(trio[,5])
for(i in 1:length(ord)){
if(trio[ord[i],2]==0 & trio[ord[i],3]==0){
ret[ord[i]] <- sample(c(0,1,2),1,prob=prob)
}else{
parents <- trio[ord[i],2:3]
pat <- ret[trio[ord[i],2]]/2
mat <- ret[trio[ord[i],3]]/2
if(pat==0.5){
pat <- sample(c(0,1),1)
}
if(mat==0.5){
mat <- sample(c(0,1),1)
}
ret[ord[i]] <- pat+mat
}
}
ret
}
make.affected.pedigree <- function(trio,p.g,r.g,h,prev){
genotype <- matrix(0,length(trio[,1]),n.loci)
for(i in 1:n.loci){
genotype[,i] <- make.genotype(trio,p.g[i,])
}
R.mat <- genotype
for(i in 1:n.loci){
tmp <- r.g[i,]
R.mat[,i] <- tmp[genotype[,i]+1]
}
R.sum <- apply(R.mat,1,sum)
var.R <- var(R.sum)
var.E <- var.R*(1-herit)/herit
E <- rnorm(length(trio[,1]),0,sqrt(var.E))
R.E <- R.sum + E
affected <- rep(0,length(trio[,1]))
affected[which(R.E>= prev)] <- 1
return(list(pedigree=trio,genotype=genotype,gen.risk.mat=R.mat,gen.risk=R.sum,risk=R.E,affected=affected))
}
library(kinship2)
plot.affected.pedigree <- function(ap){
sex <- ap$pedigree[,4]
sex[which(sex==-1)] <- 2
my.ped <- pedigree(ap$pedigree[,1],ap$pedigree[,2],ap$pedigree[,3],sex,ap$affected)
plot(my.ped,symbolosize=0.1)
}
library(igraph)
affected.pedigree <- function(pedigree,genotype,gen.risk.mat,gen.risk,risk,affected){
return(list(pedigree=pedigree,genotype=genotype,gen.risk.mat=gen.risk.mat,gen.risk=gen.risk,risk=risk,affected=affected))
}
el.from.trio <- function(trio){
non.zero <- which(apply((trio[,2:3])^2,1,sum)!=0)
el <- rbind(cbind(trio[non.zero,2],trio[non.zero,1]),cbind(trio[non.zero,3],trio[non.zero,1]))
el
}
sample.aff.sub <- function(g,aff.pedigree,k,n=NULL){
if(is.null(n)){
n <- sample(which(aff.pedigree$affected==1),1)
}
nb <- neighborhood(g,k,n,mode="all")[[1]]
ped <- matrix(aff.pedigree$pedigree[nb,],nrow=length(nb))
tmp <- ped[,2:3]
tmp[!is.element(tmp,ped[,1])] <- 0
no.parents <- which(apply(tmp,1,sum)==0)
ped[no.parents,2:3] <- 0
tmp2 <- ped[,2:3]
tobe.added <- unique(tmp2[which(!is.element(tmp2,ped[,1]))])
zero <- which(tobe.added==0)
tobe.added <- tobe.added[-zero]
nb <- c(nb,tobe.added)
ped <- aff.pedigree$pedigree[nb,]
tmp <- ped[,2:3]
tmp[!is.element(tmp,ped[,1])] <- 0
ped[,2:3] <- tmp
affected.pedigree(ped,aff.pedigree$genotype[nb,],aff.pedigree$gen.risk.mat[nb,],aff.pedigree$gen.risk[nb],aff.pedigree$risk[nb],aff.pedigree$affected[nb])
}
genotype.change <- function(g.mat){
ret <- g.mat
ret[which(ret==0)] <- "0/0"
ret[which(ret==1)] <- "0/1"
ret[which(ret==2)] <- "1/1"
ret
}
make.multi.family.table <- function(F){
family.id <- rep(1,length(F[[1]]$pedigree[,1]))
pedigree <- F[[1]]$pedigree
genotype <- genotype.change(F[[1]]$genotype)
gen.risk.mat <- F[[1]]$gen.risk.mat
gen.risk <- F[[1]]$gen.risk
risk <- F[[1]]$risk
affected <- F[[1]]$affected
if(length(F)>1){
for(i in 2:length(F)){
family.id <- c(family.id,rep(i,length(F[[i]]$pedigree[,1])))
pedigree <- rbind(pedigree,F[[i]]$pedigree)
genotype <- rbind(genotype,genotype.change(F[[i]]$genotype))
gen.risk.mat <- rbind(gen.risk.mat,F[[i]]$gen.risk.mat)
gen.risk <- c(gen.risk,F[[i]]$gen.risk)
risk <- c(risk,F[[i]]$risk)
affected <- c(affected,F[[i]]$affected)
}
}
tmp <- cbind(family.id,pedigree,affected,risk,gen.risk,gen.risk.mat,genotype)
dimnames(tmp)[[2]] <- c("family","ind","father","mother","sex","generation","affection","risk","genetic_risk",paste("locus_risk",1:length(genotype[1,]),sep="_"),paste("genotype",1:length(genotype[1,]),sep="_"))
tmp
}
n.l <- 2
g <- expand.grid(rep(list(0:2),n.l))
r.a <- c(8,2)
R <- apply(t(g) * r.a,2,sum)
age <- 0:100
Ps <- matrix(0,length(R),length(age))
for(i in 1:length(R)){
Ps[i,] <- pgamma(age,(max(R)-R[i]+7)*5,1)
}
matplot(age,t(Ps),type="l")
ps <- t(apply(Ps,1,diff))
matplot(age[-1],t(ps),type="l")
n.init <- 1
n.iter <- 10
mean.kid <-2
n.loci <- n.l
p.a <- runif(n.loci)
p.g <- cbind(p.a^2,2*p.a*(1-p.a),(1-p.a)^2)
r.g <- cbind(r.a*2,r.a,rep(0,n.loci))
herit <- 1
prev <- 0
my.ped <- my.pedigree(n.init=n.init,n.iter=n.iter,mean.kid=mean.kid)
aff.pedigree <- make.affected.pedigree(my.ped,p.g,r.g,herit,prev)
n.s <- length(my.ped[,1])
s.age <- sample(10:90,n.s,replace=TRUE)
aff <- rep(0,n.s)
R.type <- rep(0,n.s)
for(i in 1:length(R.type)){
R.type[i] <- which(R==aff.pedigree$gen.risk[i])[1]
}
for(i in 1:n.s){
if(Ps[R.type[i],s.age[i]] > runif(1)){
aff[i] <- 1
}
}
onset.age <- rep(-1,n.s)
for(i in 1:n.s){
if(aff[i]==1){
onset.age[i] <- sample(0:s.age[i],1,prob=ps[R.type[i],1:(s.age[i]+1)])
}
}
aff.pedigree$affected <- aff
plot.affected.pedigree(aff.pedigree)
library(gRain)
g.type <- paste("g",1:(length(R)),sep="")
f.type <- c("0","1")
n.g <- length(g[,1])
g.table <- g0.table <- f.table <- list()
g0.cnt <- 1
for(i in 1:n.s){
g.table[[i]] <- f.table[[i]] <- list()
}
for(i in 1:n.s){
tmp.age <- s.age[i]
tmp.p <- matrix(0,n.g,2)
for(j in 1:n.g){
tmp.p[j,2] <- Ps[j,tmp.age]
tmp.p[j,1] <- 1-tmp.p[j,2]
}
f.table[[i]] <- cptable(~p:g,values=c(t(tmp.p)),levels=f.type)
f.table[[i]]$vpa[1:2] <- paste(c("F","G"),i,sep="")
}
oya2.1 <- array(0,rep(3,3))
oya2.1[1,1,1] <- oya2.1[2,1,3] <- oya2.1[2,3,1] <- oya2.1[3,3,3] <- 1
oya2.1[1,1,2] <- oya2.1[1,2,1] <- oya2.1[2,1,2] <- oya2.1[2,2,1] <- oya2.1[2,2,2] <- oya2.1[2,2,3] <- oya2.1[2,3,2] <- oya2.1[3,2,3] <- oya2.1[3,3,2] <- 1/2
oya2.1[1,2,2] <- oya2.1[3,2,2] <- 1/4
oya2 <- array(0,rep(n.g,3))
for(i in 1:n.g){
father <- unlist(g[i,])
for(j in 1:n.g){
mother <- unlist(g[j,])
tmp.list <- list()
tmp.list[[1]] <- oya2.1[,father[1]+1,mother[1]+1]
if(n.l>1){
for(k in 2:n.l){
tmp.list[[k]] <- oya2.1[,father[k]+1,mother[k]+1]
}
}
tmp <- tmp.list[[1]]
if(n.l>1){
for(k in 2:n.l){
tmp <- outer(tmp,tmp.list[[k]],"*")
}
}
oya2[,i,j] <- c((tmp))
}
}
oya2.tandem <- c(oya2)
g <- 0:2
one.locus <- array(0,rep(3,3))
for(i in 1:3){
for(j in 1:3){
if(i==1 & j==1){
one.locus[,i,j] <- c(1,0,0)
}else if(i==1 & j==2){
one.locus[,i,j] <- c(1/2,1/2,0)
}else if(i==1 & j==3){
one.locus[,i,j] <- c(0,1,0)
}else if(i==2 & j==1){
one.locus[,i,j] <- c(1/2,1/2,0)
}else if(i==2 & j==2){
one.locus[,i,j] <- c(1/4,1/2,1/4)
}else if(i==2 & j==3){
one.locus[,i,j] <- c(0,1/2,1/2)
}else if(i==3 & j==1){
one.locus[,i,j] <- c(0,1,0)
}else if(i==3 & j==2){
one.locus[,i,j] <- c(0,1/2,1/2)
}else{
one.locus[,i,j] <- c(0,0,1)
}
}
}
g.complex <- expand.grid(rep(list(g),n.l))
n.g <- length(g.complex[,1])
oyako <- array(0,rep(n.g,3))
g.comp.pair <- expand.grid(1:n.g,1:n.g)
values <- c()
for(i in 1:length(g.comp.pair[,1])){
oya1.compg <- g.comp.pair[i,1]
oya2.compg <- g.comp.pair[i,2]
oya1.g <- unlist(g.complex[oya1.compg,])
oya2.g <- unlist(g.complex[oya2.compg,])
per.locus <- list()
for(k in 1:length(oya1.g)){
per.locus[[k]] <- one.locus[,oya1.g[k]+1,oya2.g[k]+1]
}
tmp <- as.matrix(expand.grid(per.locus),ncol=length(oya1.g))
values <- c(values,apply(tmp,1,prod))
}
gen.pop <- c(outer(p.g[1,],p.g[2,],"*"))
gen.pop <- gen.pop/sum(gen.pop)
oya2.tandem <- values
for(i in 1:n.s){
father <- mother <- 0
father <- my.ped[i,2]
if(father!=0){
father <- which(my.ped[,1]==father)
}
mother <- my.ped[i,3]
if(mother!=0){
mother <- which(my.ped[,1]==mother)
}
if(father!=0){
if(mother!=0){
g.table[[i]] <- cptable(~g:gf+gm,values=oya2.tandem,levels=g.type)
g.table[[i]]$vpa[1] <- paste("G",i,sep="")
g.table[[i]]$vpa[2] <- paste("G",father,sep="")
g.table[[i]]$vpa[3] <- paste("G",mother,sep="")
}else{
g.table[[i]] <- cptable(~g:gf+g0,values=oya2.tandem,levels=g.type)
g.table[[i]]$vpa[1] <- paste("G",i,sep="")
g.table[[i]]$vpa[2] <- paste("G",father,sep="")
g.table[[i]]$vpa[3] <- paste("G",0,i,"m",sep="")
g0.table[[g0.cnt]] <- cptable(~g,values=gen.pop,levels=g.type)
g0.table[[g0.cnt]]$vpa[1] <- paste("G",0,i,"m",sep="")
g0.cnt <- g0.cnt+1
}
}else{
if(mother!=0){
g.table[[i]] <- cptable(~g:g0+gm,values=oya2.tandem,levels=g.type)
g.table[[i]]$vpa[1] <- paste("G",i,sep="")
g.table[[i]]$vpa[2] <- paste("G",0,i,"f",sep="")
g.table[[i]]$vpa[3] <- paste("G",mother,sep="")
g0.table[[g0.cnt]] <- cptable(~g,values=gen.pop,levels=g.type)
g0.table[[g0.cnt]]$vpa[1] <- paste("G",0,i,"f",sep="")
g0.cnt <- g0.cnt+1
}else{
g.table[[i]] <- cptable(~g:g0+g02,values=oya2.tandem,levels=g.type)
g.table[[i]]$vpa[1] <- paste("G",i,sep="")
g.table[[i]]$vpa[2] <- paste("G",0,i,"f",sep="")
g.table[[i]]$vpa[3] <- paste("G",0,i,"m",sep="")
g0.table[[g0.cnt]] <- cptable(~g,values=gen.pop,levels=g.type)
g0.table[[g0.cnt]]$vpa[1] <- paste("G",0,i,"m",sep="")
g0.cnt <- g0.cnt+1
g0.table[[g0.cnt]] <- cptable(~g,values=gen.pop,levels=g.type)
g0.table[[g0.cnt]]$vpa[1] <- paste("G",0,i,"f",sep="")
g0.cnt <- g0.cnt+1
}
}
}
gf.list <- list()
for(i in 1:length(g.table)){
gf.list[[i]] <- g.table[[i]]
}
cnt <- length(gf.list)
for(i in 1:length(g0.table)){
gf.list[[cnt+i]] <- g0.table[[i]]
}
cnt <- length(gf.list)
for(i in 1:length(f.table)){
gf.list[[cnt+i]] <- f.table[[i]]
}
cptlist <- compileCPT(gf.list)
pn <- grain(cptlist)
pn
summary(pn)
plot(pn)
gen.pop
querygrain(pn)
querygrain(pn,nodes=paste("G",0,sep=""))
querygrain(pn,nodes=paste("G",0,1,sep=""))
querygrain(pn,nodes=paste("G",1:n.s,sep=""))
pn2 <- setEvidence(pn,paste("G",0,sep=""),paste("g",1,sep=""))
pn3 <- setEvidence(pn2,paste("G",0,1,sep=""),paste("g",2,sep=""))
querygrain(pn3)
ev.1 <- ev.2 <- c()
for(i in 1:n.s){
ev.1 <- c(ev.1,paste("F",i,sep=""))
ev.2 <- c(ev.2,as.character(aff.pedigree$aff[i]))
}
pn.F <- setEvidence(pn, ev.1,ev.2)
querygrain(pn.F,nodes=c("G1"))
querygrain(pn,nodes=c("G1"))
g.available <- sample(2:n.s,5)
ev.g.1 <- ev.g.2 <- c()
for(i in 1:length(g.available)){
ev.g.1 <- c(ev.g.1,paste("G",g.available[i],sep=""))
tmp.g <- aff.pedigree$genotype[g.available[i],]
tmp.g.2 <- sum((tmp.g)*3^((length(tmp.g)-1):0))+1
ev.g.2 <- c(ev.g.2,paste("g",tmp.g.2,sep=""))
}
pn.F.G <- setEvidence(pn.F,ev.g.1,ev.g.2)
querygrain(pn.F.G,nodes=c("G1"))
querygrain(pn.F,nodes=c("G1"))
querygrain(pn,nodes=c("G1"))
