n <- 1000
prob <- c(0.5,0.5)
censor <- sample(0:1,n,replace=TRUE,prob=prob)
times <- sort(rexp(n))
x <- ((n-1):0)/(n:1)
v <- rep(1,length(x))
v[which(censor == 1)] <- x[which(censor ==1)]
Data <- data.frame(Censor = censor, Time = times)
library(survival)
KMcurve <- survfit(Surv(Time, Censor)~1, data=Data)
plot(KMcurve, conf.int=FALSE, mark.time=TRUE)
n1 <- 1000
n2 <- 1000
p1 <- 1
p2s <- seq(from=1,to=2,length=9)
n.iter <- 1000
cross <- matrix(0,length(p2s),n.iter)
res <- list()
for(j in 1:length(p2s)){
p2 <- p2s[j]
res[[j]] <- array(0,c(n.iter,n1+1,2))
for(i in 1:n.iter){
x1 <- cumsum(rexp(n1,p1))
x2 <- cumsum(rexp(n2,p2))
res[[j]][i,,1] <- c(0,x1)
res[[j]][i,,2] <- c(0,x2)
time1 <- quantile(x1,0.05)
time2 <- quantile(x1,0.1)
x1.1 <- length(which(x1>time1))/n1
x1.2 <- length(which(x1>time2))/n1
x2.1 <- length(which(x2>time1))/n2
x2.2 <- length(which(x2>time2))/n2
if((x1.1-x2.1)*(x1.2-x2.2)<0){
cross[j,i] <- 1
}
}
}
num.frac <- apply(cross,1,sum)/n.iter
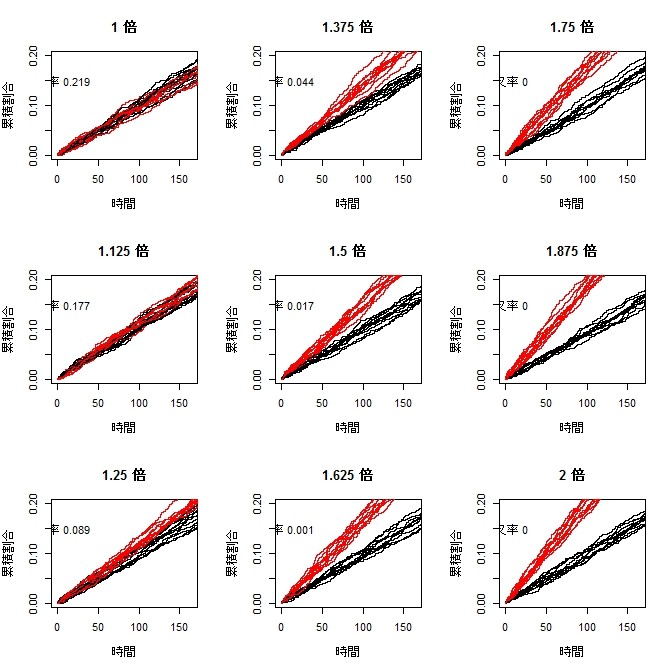
par(mfcol=c(3,3))
for(j in 1:length(p2s)){
plot(res[[j]][1,,1],(0:n1)/n1,type="s",xlim=c(0,quantile(x1,0.15)),ylim=c(0,0.2),xlab="時間",ylab="累積割合",main=paste(p2s[j]/p1,"倍"))
points(res[[j]][1,,2],(0:n2)/n2,type="s",col=2)
for(i in 2:10){
points(res[[j]][i,,1],(0:n1)/n1,type="s")
points(res[[j]][i,,2],(0:n2)/n2,type="s",col=2)
}
text(0.05,0.15,paste("交叉率",num.frac[j]))
}
par(mfcol=c(1,1))
Data <- data.frame(Censor = c(cens1,cens2), Time = c(x1,x2), Drug = c(rep(1,n1),rep(2,n2)))
surv.out <- survfit(Surv(Data$Time,Data$Censor)~strata(Data$Drug))
plot(surv.out)
