- ネットワークに介入する(遺伝影響)
- 遺伝子多型などの影響は、因子(分子)の機能に多少の多寡が生じたり、ネットワークを通じて発現促進・抑制をする効率が変化したりする。これは、推移行列の値(微分方程式の係数)に違いをもたらす
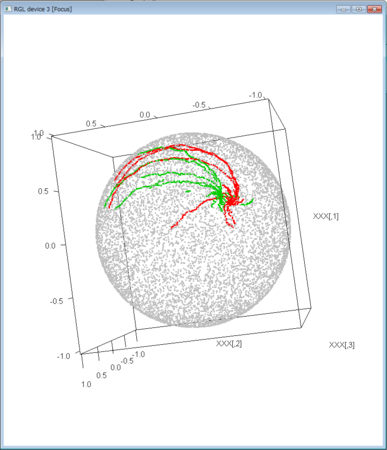
- 以下のソースでは、推移行列をわずかな角度で回転させ、推移行列に少しの違いを生じさせた上で、どのような状態変化が起きるかを示した
- 比較的安定しているときの複数因子の量の分布に違いがあるし、大きく揺らいだあとの、複数因子の収束の道筋が異なることもわかる
n<-3
M<-Random.Start(n)
Nrep<-4
Niter<-1000
plotstart<-1
commondrift<-0.005
raredrift<-0.1
rarefreq<-0.005
dt<-0.01
dt2<-0.2
S<-diag(n)
S1<-matrix(c(cos(dt),sin(dt),-sin(dt),cos(dt)),2,2)
S[1:2,1:2]<-S1
SX1<-diag(n)
SR1<-matrix(c(cos(dt2),sin(dt2),-sin(dt2),cos(dt2)),2,2)
SX1[1:2,1:2]<-SR1
SR2<-matrix(c(cos(-dt2),sin(-dt2),-sin(-dt2),cos(-dt2)),2,2)
SX2<-diag(n)
SX2[1:2,1:2]<-SR2
M2<-M%*%SX2
M<-M%*%SX1
Xsum<-NULL
tmpcol<-c()
currentM<-diag(n)
for(ir in 1:Nrep){
X<-matrix(0,Niter,n)
X[1,]<-runif(n)
X[1,]<-X[1,]/sqrt(sum(X[1,]^2))
for(i in 2:Niter){
P<-X[i-1,]
Q<-M%*%P
PQ<-Q-P
R<-XYRotation(P,PQ)
tmpP<-R%*%P
tmpQ<-S%*%tmpP
newQ<-solve(R)%*%tmpQ
currentdrift<-commondrift
if(runif(1)<rarefreq)currentdrift<-raredrift
X[i,]<-newQ+rnorm(n)*currentdrift
X[i,]<-X[i,]/sqrt(sum(X[i,]^2))
}
Xsum<-rbind(Xsum,X[plotstart:length(X[,1]),])
tmpcol<-c(tmpcol,rep(ir,(Niter-plotstart+1)))
}
normX<-sqrt(sum(Xsum[1,]^2))
NrandPt<-10000
XX<-matrix(rnorm(NrandPt*n),NrandPt,n)
XX<-XX/sqrt(apply(XX^2,1,sum))*normX
XXX<-rbind(XX,Xsum)
col<-c(rep("gray",length(XX[,1])),tmpcol)
plot3d(XXX,col=col)
lims<-range(Xsum)
plot3d(Xsum,col=tmpcol,xlim=lims,ylim=lims,zlim=lims)
XsumOri<-Xsum
tmpcolOri<-tmpcol
Mori<-M
M<-M2
Xsum<-NULL
tmpcol<-c()
currentM<-diag(n)
for(ir in 1:Nrep){
X<-matrix(0,Niter,n)
X[1,]<-runif(n)
X[1,]<-X[1,]/sqrt(sum(X[1,]^2))
for(i in 2:Niter){
P<-X[i-1,]
Q<-M%*%P
PQ<-Q-P
R<-XYRotation(P,PQ)
tmpP<-R%*%P
tmpQ<-S%*%tmpP
newQ<-solve(R)%*%tmpQ
currentdrift<-commondrift
if(runif(1)<rarefreq)currentdrift<-raredrift
X[i,]<-newQ+rnorm(n)*currentdrift
X[i,]<-X[i,]/sqrt(sum(X[i,]^2))
}
Xsum<-rbind(Xsum,X[plotstart:length(X[,1]),])
tmpcol<-c(tmpcol,rep(ir,(Niter-plotstart+1)))
}
normX<-sqrt(sum(Xsum[1,]^2))
NrandPt<-10000
XX<-matrix(rnorm(NrandPt*n),NrandPt,n)
XX<-XX/sqrt(apply(XX^2,1,sum))*normX
XXX<-rbind(XX,Xsum)
col<-c(rep("gray",length(XX[,1])),tmpcol)
plot3d(XXX,col=col)
lims<-range(Xsum)
plot3d(Xsum,col=tmpcol,xlim=lims,ylim=lims,zlim=lims)
X1sumX2sum<-rbind(XsumOri,Xsum)
XXX<-rbind(XX,X1sumX2sum)
col<-c(rep("gray",length(XX[,1])),rep(2,length(tmpcolOri)),rep(3,length(tmpcol)))
plot3d(XXX,col=col)
plot3d(X1sumX2sum,col=c(rep(2,length(tmpcolOri)),rep(3,length(tmpcol))),xlim=lims,ylim=lims,zlim=lims)
library(GPArotation)
Cartesian2AngularX<-function(x){
n<-length(x)
if(n==1){
return(list(r=abs(x),t=acos(sign(x))))
}else if(n==2){
tmpt<-0
if(x[1]!=0){
tmpt<-acos(x[1]/sqrt(sum(x^2)))*sign(x[2])
if(sign(x[2])==0){
if(x[1]>0){
tmpt<-0
}else if(x[1]<0){
tmpt<-pi
}
}
}else{
if(x[2]>0){
tmpt<-pi/2
}else if(x[2]<0){
tmpt<--pi/2
}
}
return(list(r=sqrt(sum(x^2)),t=tmpt))
}
r<-sqrt(sum(x^2))
xst<-x/r
t<-rep(0,n-1)
S<-C<-t
S[n-1]<-xst[n]
if(S[n-1]>1)S[n-1]<-1
if(S[n-1]<(-1))S[n-1]<-(-1)
t[n-1]<-asin(S[n-1])
C[n-1]<-cos(t[n-1])
cumC<-C[n-1]
for(i in (n-2):1){
if(cumC!=0){
S[i]<-xst[i+1]/cumC
if(S[i]>1)S[i]<-1
if(S[i]<(-1))S[i]<-(-1)
t[i]<-asin(S[i])
C[i]<-cos(t[i])
cumC<-cumC*C[i]
}else{
S[i]<-0
t[i]<-asin(S[i])
C[i]<-cos(t[i])
}
}
list(r=r,t=t)
}
VectorRotation<-function(P,inv=FALSE){
n<-length(P)
if(n==1){
return(matrix(c(1),1,1))
}
Q<-Cartesian2AngularX(P)
M<-diag(n)
M2<-M
for(i in 1:length(Q[[2]])){
tmp<-M
tmp[i,i]<--sin(Q[[2]][i])
tmp[i,i+1]<-cos(Q[[2]][i])
tmp[i+1,i]<-cos(Q[[2]][i])
tmp[i+1,i+1]<-sin(Q[[2]][i])
M2<-tmp%*%M2
}
M2<-t(M2)[,n:1]
S<-sign(M2[,1]*P)
M2<-M2*S
if(inv){
M2<-solve(M2)
}
M2
}
XYRotation<-function(P,V,inv=TRUE){
R1<-VectorRotation(P,inv=TRUE)
V2<-R1%*%V
tmpV<-V2[2:length(V)]
tmpR2<-VectorRotation(tmpV,inv=TRUE)
R2<-diag(length(V))
R2[2:n,2:n]<-tmpR2
ret<-R2%*%R1
if(!inv)ret<-solve(ret)
ret
}
