
- nature_biotechnology論文
- bioconductorサイト
- cytoSPADEのステップ
- 1. Density-dependent down-sampling
- 細胞ごとに、局所濃度推定値を出す(k-NN濃度でもよさそう)
- 細胞間距離はL1距離(マンハッタン距離・ハミング距離のことだろう)を使う
- 最近接点距離の分布をとり、その中央値(med_min_dist)を求める(中央値なので、全細胞間距離を取らずにランダムサンプリングして2000個の細胞の最近接点距離を測る)
- med_min_distの定数倍(defaultで5)を、近傍閾値とする(近傍閾値以内に最近接点を持つ割合をコントロールするように、最近接点距離分布から閾値を定めるのも手だろう)
- 近傍閾値内に含まれる点の数をその細胞の位置での局所濃度推定値とする
- 濃度依存ダウンサンプリングのための濃度閾値を定める
- 二つの濃度閾値を用いて、以下に示すようにダウンサンプリングする
- その閾値を全細胞の局所濃度推定値のクオンタイル値で定める、1%をOD、3%をTDとする
- ここの処理をランダムサンプリングした細胞での局所濃度推定値のクオンタイルで代用すると処理が軽そうだが…
- 細胞の局所濃度推定値をOD,TDに照らしてサンプリング確率を決める
- OD以下の濃度であれば、サンプリングしない
- ODより濃度が大きく、TD以下であれば、必ずサンプリングする
- TDより濃度が大きければ、濃度に反比例するように(濃ければ濃いほどサンプリング確率を小さく)サンプリングする。ただし、ちょうどTDのときには必ずサンプリングすることで、ODより大、LD以下の場合と連続性を持たせる
- ここまでをRでやってみよう
library(vegan)
library(GPArotation)
library(igraph)
library(rgl)
library(MCMCpack)
vision.scale <- function(x,scl){
(x-min(x))/(max(x)-min(x)) * scl + (1-scl)/2
}
vision.standard <- function(x,scl = 0.9){
rg <- apply(x,2,range)
tmp <- apply(x,2,vision.scale, scl)
return(list(st.x = tmp,rg=rg))
}
round.coords <- function(x,n.cell=50){
d <- length(x[1,])
matrix(floor(x * n.cell),ncol=d)
}
library(vegan)
library(GPArotation)
library(igraph)
library(rgl)
library(MCMCpack)
my.FACS.pts <- function(d,n.steps=10,n.pt.range=100:1000,NN=100,min.v=5,max.v=10,v=0.3){
X <- matrix(0,0,d)
tmp.ns <- sample(n.pt.range,n.steps,replace=TRUE)
tmp.Ls <- runif(n.steps,min.v,max.v)
for(i in 1:n.steps){
tmp.n <- tmp.ns[i]
tmp.X <- matrix(0,tmp.n,d)
tmp.L <- tmp.Ls[i]
tmp.V <- seq(from=0,to=1,length=tmp.n*NN)
tmp.prob <- (tmp.V-mean(tmp.V))^2
tmp.prob <- tmp.prob/sum(tmp.prob)
tmp.S <- tmp.V[sort(sample(1:length(tmp.V),tmp.n,replace=TRUE,prob=tmp.prob))]*tmp.L
tmp.X[,1] <- tmp.S
tmp.R <- Random.Start(d)
tmp.X <- tmp.X %*% tmp.R
if(i > 1){
tmp.st <- sample(1:length(X[,1]),1)
tmp.st.2 <- 1
st.pt <- X[tmp.st,]
connect.pt <- tmp.X[tmp.st.2,]
tmp.X <- t(t(tmp.X) + st.pt - connect.pt)
}
X <- rbind(X,tmp.X)
}
X <- X + rnorm(length(X),0,v)
X.curve <- X
for(i in 1:d){
X.curve[,i] <- sign(X.curve[,i]) * abs(X.curve[,i]^2)
}
X <- X.curve
return(X)
}
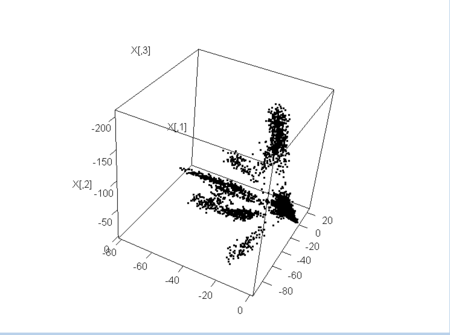
d <- 3
n.steps <- 10
n.pt.range <- 100:1000
NN <- 100
my.X <- my.FACS.pts(d,n.steps,n.pt.range,NN)
plot(as.data.frame(my.X))
vision.scale <- function(x,scl){
(x-min(x))/(max(x)-min(x)) * scl + (1-scl)/2
}
vision.standard <- function(x,scl = 0.9){
rg <- apply(x,2,range)
tmp <- apply(x,2,vision.scale, scl)
return(list(st.x = tmp,rg=rg))
}
round.coords <- function(x,n.cell=50){
d <- length(x[1,])
matrix(floor(x * n.cell),ncol=d)
}
d <- 3
n.steps <- 10
n.pt.range <- 100:1000
NN <- 100
my.X <- my.FACS.pts(d,n.steps,n.pt.range,NN)
plot(as.data.frame(my.X))
plot3d(my.X[,1:3])
-
-
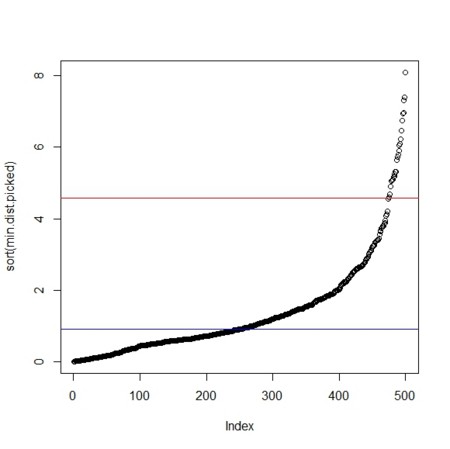
- 点をサンプリングして最近点距離分布をとる(ソートしてプロット)。その中央値と中央値のα=5倍を示しておく
n <- length(X[,1])
print(n)
n.pick <- 500
picked <- sample(1:n,n.pick)
min.dist.picked <- rep(0,n.pick)
for(i in 1:n.pick){
tmp <- t(X)-X[picked[i],]
tmp2 <- apply(abs(tmp),2,sum)
tmp2[which(tmp2==0)] <- max(tmp2)+1
min.dist.picked[i] <- min(tmp2)
}
plot(sort(min.dist.picked))
mdan <- median(min.dist.picked)
abline(h=mdan,col=4)
alpha <- 5
L <- mdan*alpha
abline(h=L,col=2)
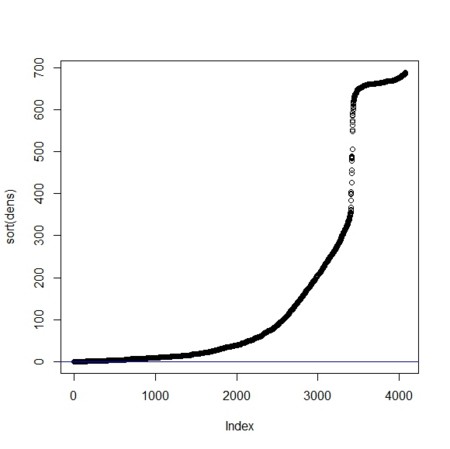
dens <- rep(0,n)
for(i in 1:n){
tmp <- t(X)-X[i,]
tmp2 <- apply(abs(tmp),2,sum)
tmp2[which(tmp2==0)] <- max(tmp2)+1
dens[i] <- length(which(tmp2<=L))
}
OD.perc <- 0.01
TD.perc <- 0.03
OD.TD <- quantile(dens,c(OD.perc,TD.perc))
plot(sort(dens))
abline(h=OD.TD[1],col=2)
abline(h=OD.TD[2],col=4)

dwn.smpl <- rep(0,n)
for(i in 1:n){
if(dens[i] <=OD.TD[1]){
}else if(dens[i] <= OD.TD[2]){
dwn.smpl[i] <- 1
}else{
if(OD.TD[2]==0){
tmp.prob <- 1/dens[i]
}else{
tmp.prob <- OD.TD[2]/dens[i]
}
dwn.smpl[i] <- sample(0:1,1,prob=c(1-tmp.prob,tmp.prob))
}
}
sum(dwn.smpl)/n
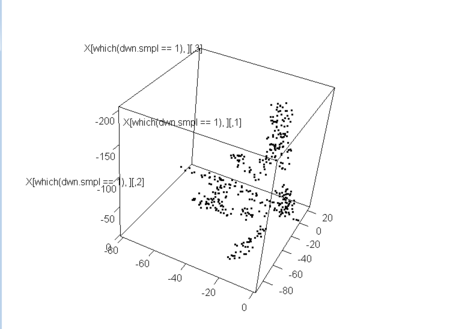
X.dwn <- X[which(dwn.smpl==1),]
plot3d(X.dwn)
- 2. クラスターに分ける
- ダウンサンプリングしてあるので距離行列の計算も楽だし、クラスタリングも楽
- 距離はハミング距離
- クラスタリングは凝集法でhclust(method="single")
- 指定クラスタ数に分けるのにcutree()を使う
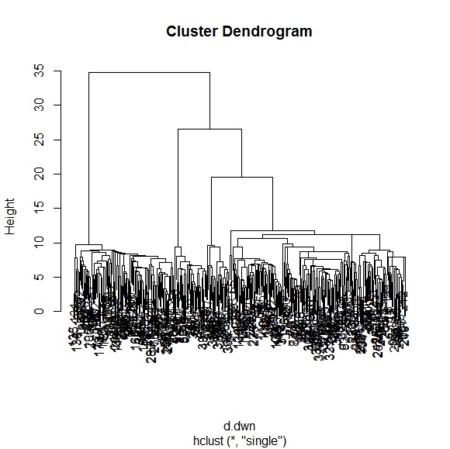
d.dwn <- dist(X.dwn,method="manhattan")
cl <- hclust(d.dwn,method="single")
num.cl <- 5
cl2 <- cutree(cl, k=num.cl)
cl.coords <- matrix(0,num.cl,length(X.dwn[1,]))
for(i in 1:num.cl){
tmp <- X.dwn[which(cl2==i),]
if(is.matrix(tmp)){
cl.coords[i,] <- apply(tmp,2,median)
}else{
cl.coords[i,] <- tmp
}
}
-
- 各クラスタを単ノードのサブグラフとみなし、サブグラフをつなぐべくエッジを1本ずつ加えている。サブグラフをランダムに選び、最短距離にあるサブグラフとつなぐ。最短距離とは、サブグラフ上のノードとサブグラフ上でないノードの距離のすべてのうちで最短のもの(Boruvkaアルゴリズム)
- ここではnnclustパッケージのmst()を使おう
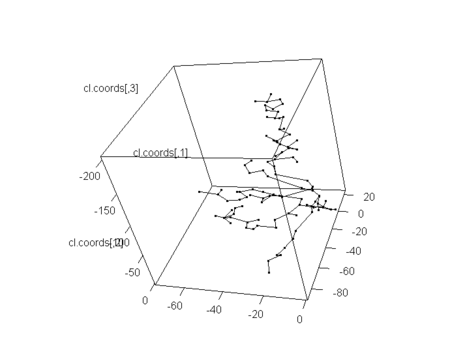
library(nnclust)
a<-mst(cl.coords)
plot(cl.coords[,1:2])
segments(cl.coords[a$from,1], cl.coords[a$from,2], cl.coords[a$to,1],
cl.coords[a$to,2],col="blue")
plot3d(cl.coords)
for(i in 1:length(a$from)){
seg.m <- matrix(c(cl.coords[a$from[i],1],cl.coords[a$from[i],2],cl.coords[a$from[i],3],cl.coords[a$to[i],1],cl.coords[a$to[i],2],cl.coords[a$to[i],3]),byrow=TRUE,nrow=2)
segments3d(seg.m)
}
- 4 全細胞を木構造につなげるべくup-samplingする
- すべての点について、ダウンサンプルの中で最も近い点が属するクラスタのメンバーとする
- 下の図では、そのようにして帰属させた点の体積が帰属する点の数に比例するようにプロットしたもの

n.members <- rep(0,num.cl)
for(i in 1:n){
tmp <- t(X.dwn)-X[i,]
tmp2 <- apply(abs(tmp),2,sum)
tmp3 <- which(tmp2==min(tmp2))[1]
tmp4 <- cl2[tmp3]
n.members[tmp4] <- n.members[tmp4] + 1
}
plot3d(cl.coords)
for(i in 1:length(a$from)){
seg.m <- matrix(c(cl.coords[a$from[i],1],cl.coords[a$from[i],2],cl.coords[a$from[i],3],cl.coords[a$to[i],1],cl.coords[a$to[i],2],cl.coords[a$to[i],3]),byrow=TRUE,nrow=2)
segments3d(seg.m)
}
max.r <- max(cl.coords)-min(cl.coords)
for(i in 1:num.cl){
spheres3d(cl.coords[i,],radius=(n.members[i]/max(n.members))^(1/length(X[1,]))*max.r/20)
}







