- Rもpythonもjupyter notebookで文書作成できる
- 3次元プロットをしなくてよいのなら、Rでは普通の二次元プロッティング関数を使えばよいし、Pythonではmatplotlibを使えばよい
- 3次元プロットを描いてぐりぐり動かすとなると、Rではplot3dを使い、pythonでは…、plotlyが使えるか…となる
- が、両方を別々に覚えるのが、記憶力の減退と共につらい
- jupyter notebook上ならRもPythonもplotlyで3Dぐりぐりができるということらしいので、両者を比較しながら、使ってみることにする
- 準備
install.packages("plotly")
library(plotly)
pip install plotly
-
- が必要なのは陶然として、jupyter内で動かすには
pip install npm
jupyter labextension install @jupyterlab/plotly-extension
-
- としないと、plotlyの画像出力がjupyter notebook上に現れない
import plotly.offline as offline
import plotly.graph_objs as go
offline.init_notebook_mode()
-
- として、ようやく、offline.iplot()関数にpyplotで作ったグラフ表示情報オブジェクトを渡すことで、グラフが現れる
- 散布図
p <- seq(from=0,to=1,length=100) * 10
q <- sin(p)
fig = plot_ly(x=p,y=q,type="scatter")
fig
-
- Pythonの場合
- もともとのplotlyはplotly.graph_objsという仕様で、細かく描画条件を指定するようになっているらしいが、さすがに面倒だ、ということで、簡単にした(機能を落としつつ、コマンドを簡略化した)plotly.expressというものがある。以下ではそれを使う
import numpy
import plotly.express as px
p = numpy.arange(100) * 0.1
q = numpy.sin(p)
fig = px.scatter(x=p, y=q)
fig.show()
-
- ちなみにこのplotly.expressはplotly.graph_objects.Figureを返すので、fig.show() として描画することも、offline.iplot()として描画することもできるようである
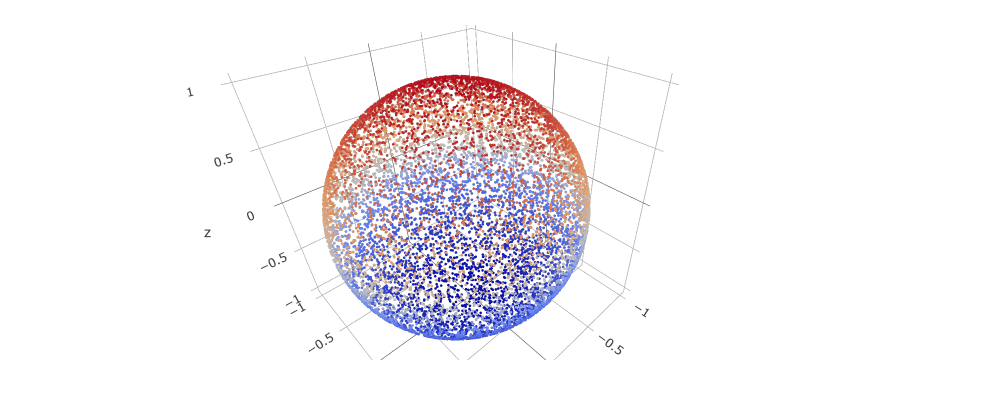
- 3D scatter plot
- Rの場合(このブログでは動かせないがjupyter notebook上では動かせる)
xyz <- matrix(rnorm(10000*3),ncol=3)
xyz <- xyz/sqrt(apply(xyz^2,1,sum))
fig2 <- plot_ly(x=xyz[,1],y=xyz[,2],z=xyz[,3],type="scatter3d",marker=list(size=1,color=xyz[,3]))
fig2
-
- Pythonの場合
- 2D scatterplotではplotly.expressを使ったが、3D scatterplotだと、点のサイズ指定で(少なくとも自分の環境では)エラーがでるので、簡易版expressではなく、plotly.graph_objsを使うこととする
import plotly.offline as offline
import plotly.graph_objs as go
offline.init_notebook_mode()
import numpy
import numpy.random
def my_unit_vector(X):
return(X/numpy.linalg.norm(X,axis=1).reshape(-1,1))
xyz = numpy.array(numpy.random.randn(1000,3))
xyz_ = my_unit_vector(xyz)
trace = go.Scatter3d(
x = xyz_[:,0], y = xyz_[:,1], z = xyz_[:,2],mode = 'markers', marker = dict(
size = 1)
)
fig = go.Figure(data = [trace])
offline.iplot(fig)
-
- 以下のように、描くべきオブジェクト fig を作って、offline.plot()に渡すだけでも描ける
fig = go.Figure(go.Scatter3d(x = xyz_[:,0], y = xyz_[:,1], z = xyz_[:,2],mode = 'markers', marker = dict(size = 1)))
offline.iplot(fig)
-
- 最後の描画のところは、offline.iplot(fig)の代わりに
fig.show()
-
- でも3Dぐりぐりプロットが現れる(ようだ)
- もう、細かいことはいいから、Rのplot3d()的に描ければいいや、ということなら、関数を定義して
def my_go_scatter3d(X,size=1):
fig = go.Figure(go.Scatter3d(x =X[:,0], y = X[:,1], z = X[:,2],mode = 'markers', marker = dict(size = size)))
offline.iplot(fig)
my_go_scatter3d(xyz_)
-
- pythonのnumpy.apply_along_axis()関数
def my_unit_vector(X):
return(X/numpy.linalg.norm(X,axis=1).reshape(-1,1))
-
- という関数を定義して使った。これは、numpyの線形代数関連関数を納めたlinalg配下にあるnormを取る関数を列(axis=1)に関してつぶした結果を返すという処理。この処理は(おそらく)高速実装されている。
- これに似たやり方で、自作の関数 my_fun()を定義して、同様に、numpy ndarrayに作用するには
numpy.apply_along_axis(my_fun,axis=1,m)
-
- のようにして、ndarray オブジェクトm(行列と仮定しておく)の列(axis=1)をつぶすようにmy_fun()を各行に処理させることができる
- ただし、これは、ループ処理を簡略化して書いてあるだけなので、mの各要素にアクセスするそうで、速くなるわけではないらしい(Rのapply()関数は、場合によっては、ベクトル演算化できるところはベクトル演算化して速くしてくれたはずだが…。要するに、Rで言えばlapply()関数的なものということなのだろう)
- それよりは、ベクトル化した(必要ならループを使った)コードを書く方が速いらしい(参考はこちら)
- Python の plotlyは、pythonのディクショナリ形式で必要な情報を持たせて、それをplotly.graph_objects.Figure 形式に修正してFigureデータタイプで持たせ、その情報を読んで視覚化する、という仕組み:こちら。
- それが解れば、あとは、例(https://plotly.com/python/:tilte=こちら)を参考に、合わせて行けばよいようだ


