- ネットワークの因子たち(ロトカ=ヴォルテラ編)
- 多くの分子はネットワークを形成し、フィードバック等の制御構造がある
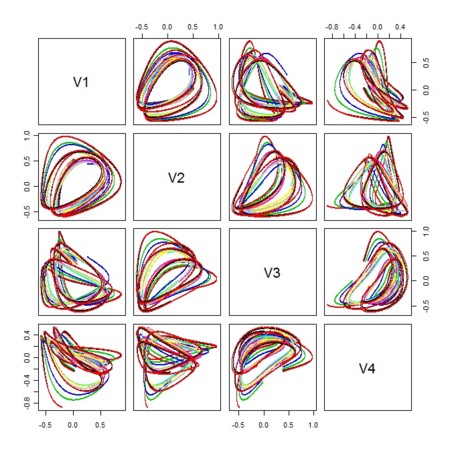
- 単純な構造として、ロトカ=ヴォルテラ(たとえばこちら)のような反応ループ関係を取り上げる
- 初期値が異なるだけで、ループの構成因子の濃度分布は異なってくる
- 個体差はこんなところにもあり、ジェノタイプによって、ループ構成因子の(ある時点の)濃度が定まれば、その後の周期的変動には個体差があり、それは、個々の因子の濃度の平均・分散・最大・最小などに影響を及ぼす
library(sphere)
library(rgl)
StandardLotkaVolterra20111005<-function(n=4,Nrep=30,drift=FALSE,driftk=0.05,Niter=1000,dt=0.01,us=NULL){
if(is.null(us)){
us<-rep(1,n)
}
Nr<-n
ks<-runif(Nr)
ks<-rep(1,n)
In<-Player<-diag(n)
Out<-matrix(0,Nr,n)
for(i in 1:Nr){
for(j in 1:n){
if(j==i+1){
Player[i,j]<-1
Out[i,j]<-1
}
}
}
Player[Nr,1]<-Out[Nr,1]<-1
S<-1
P<-runif(1,min=0,max=(1/n)^n)
xssum<-NULL
col<-c()
m<-matrix(Niter,n)
for(rep in 1:Nrep){
if(drift){
if(rep>1){
prevm<-m
m<-prevm
p<-rep(0,Niter)
p[1]<-0
for(i in 1:n){
p[1]<-p[1]+us[i]*m[1,i]
}
m[1,]<-m[1,]/p[1]
p[1]<-0
for(i in 1:n){
p[1]<-p[1]+us[i]*m[1,i]
}
m[1,]<-prevm[1,]+abs(rnorm(n)*driftk)
}else{
m<-matrix(0,Niter,n)
p<-rep(0,Niter)
p[1]<-0
for(i in 1:n){
p[1]<-p[1]+us[i]*m[1,i]
}
m[1,]<-m[1,]/p[1]
p[1]<-0
for(i in 1:n){
p[1]<-p[1]+us[i]*m[1,i]
}
m[1,]<-runif(n)
}
}else{
m<-matrix(0,Niter,n)
p<-rep(0,Niter)
p[1]<-0
for(i in 1:n){
p[1]<-p[1]+us[i]*m[1,i]
}
m[1,]<-m[1,]/p[1]
p[1]<-0
for(i in 1:n){
p[1]<-p[1]+us[i]*m[1,i]
}
m[1,]<-runif(n)
}
library(MCMCpack)
for(i in 2:Niter){
m[i,]<-m[i-1,]
for(j in 1:Nr){
tmp<-ks[j]*dt
pamountIn<-0
pamountOut<-0
for(k in 1:n){
tmp<-tmp*m[i-1,k]^Player[j,k]
pamountIn<-pamountIn+us[k]*In[j,k]
pamountOut<-pamountOut+us[k]*Out[j,k]
}
for(k in 1:n){
m[i,k]<-m[i,k]-In[j,k]*tmp+tmp*pamountIn/pamountOut*us[k]*Out[j,k]/pamountOut*Out[j,k]
}
}
for(j in 1:n){
p[i]<-p[i]+us[j]*m[i,j]
}
}
xs<-matrix(0,Niter,n-1)
cv<-CategoryVector(n)
for(i in 1:Niter){
for(j in 1:n){
xs[i,]<-xs[i,]+m[i,j]*cv[j,]
}
}
xssum<-rbind(xssum,xs)
col<-c(col,rep(rep,Niter))
}
plot3d(xssum[,1],xssum[,2],xssum[,3],col=col)
list(X=xssum,col=col,In=In,Player=Player,Out=Out,m=m)
}
LV.out<-StandardLotkaVolterra20111005(n=5,Nrep=10,drift=TRUE)
plot(as.data.frame(LV.out[[1]]),col=LV.out[[2]],cex=0.01)
