- こちらで、ランダムグラフを勉強している
- Y染色体の伝搬をランダムグラフで扱ったり、巨大な連結グラフに確率過程を持ち込むことで、数学的に扱って、グラフの特徴を定性的・定量的に求める考え方が書かれている
- 染色体の伝搬をY染色体のように単純に考えずに組換えありで扱うと、どんな風になるか(扱えるのかも含めて)、夏休みらしく、うまく行かなくても、いいか、というスタンスでやってみることにする
- 変数設定
- グラフの構成成分
- ノードは、すべての塩基とする
- エッジは、染色体上のつながりと、親子関係の伝達との2種類とする
- ノードの属性
- ノードは座標を持つ
- 染色体をこの世という空間にある点とみなしたときの、その位置座標
- 染色体上の塩基番号という座標
- 染色体には、必ず交叉が起きるというルールがあるので、染色体に必ず交叉を入れるような設定にして、染色体間の距離は、そのような交叉発生のルールのもとで、ほぼ0.5の確率で奇数回の交叉が入るようなものを取ればよさそうだ
- 時刻という座標
- エッジの属性
- 染色体というつながりか、伝達というつながりか、の2種類に分かれる
- そうは言っても
- まず、染色体のグラフを作って、その上で、塩基のノードとそれらの関連にエッジをつけるのが手順としてはよさそうだ
- まずはイザナギ・イザナミ(アダムとイブ)からスタートにしよう
- 世代ごとに少しずつ人口をふやしつつ
- ペアリングに制約を入れることを考慮し
- 移住しながら…
init.n <- 2
n.gen <- 50
pop.gen <- c(init.n)
loc <- list()
loc[[1]] <- matrix(0,pop.gen[1],2)
loc[[1]] <- loc[[1]] + rnorm(length(loc[[1]]))
parents <- list()
parents[[1]] <- matrix(0,pop.gen[1],2)
thres = 0
incr.ratio <- 1.05
migration <- runif(2)
for(i in 2:n.gen){
d <- matrix(0,pop.gen[i-1],pop.gen[i-1])
for(j in 1:(pop.gen[i-1]-1)){
for(j2 in (j+1):pop.gen[i-1]){
d[j,j2] <- d[j2,j] <- sum((loc[[i-1]][j,]-loc[[i-1]][j2,])^2)
}
}
d[which(d <= thres)] <- Inf
prob <- exp(-d^2/2)
prob <- prob/sum(prob)
max.child <- pop.gen[i-1]*incr.ratio * 5
n.child <- sample(2:max.child,1,prob = dpois(2:max.child,pop.gen[i-1]*incr.ratio))
pop.gen <- c(pop.gen, n.child)
parents.number <- sample(1:length(d),pop.gen[i],replace=TRUE,prob = prob)
address <- matrix(1:length(d),pop.gen[i-1],pop.gen[i-1])
parents.list <- which(address <= max(address),arr.ind=TRUE)
parents[[i]] <- parents.list[parents.number,]
loc[[i]] <- (loc[[i-1]][parents[[i]][,1],] + loc[[i-1]][parents[[i]][,2],])/2
loc[[i]] <- loc[[i]] + cbind(rnorm(length(loc[[i]][,1]),rnorm(1,migration[1])),rnorm(length(loc[[i]][,2]),rnorm(1,migration[2])))
}
xlim <- ylim <- range(unlist(loc))
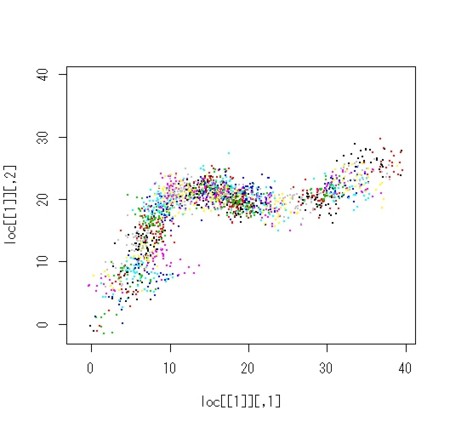
plot(loc[[1]],xlim = xlim, ylim = ylim,col=1,cex = 0.5,pch =20)
for(i in 2:n.gen){
points(loc[[i]],col=i,cex = 0.5,pch=20)
Sys.sleep(0.1)
}
library(MCMCpack)
init.n <- 10
n.gen <- 100
pop.gen <- c(init.n)
loc <- list()
loc[[1]] <- matrix(0,pop.gen[1],2)
loc[[1]] <- loc[[1]] + rnorm(length(loc[[1]]),sd=1)
parents <- list()
parents[[1]] <- matrix(0,pop.gen[1],2)
thres = mean(abs((c(loc[[1]]))))
incr.ratio <- 1.01
migration <- runif(2)
for(i in 2:n.gen){
d <- matrix(0,pop.gen[i-1],pop.gen[i-1])
for(j in 1:(pop.gen[i-1]-1)){
for(j2 in (j+1):pop.gen[i-1]){
d[j,j2] <- d[j2,j] <- sum((loc[[i-1]][j,]-loc[[i-1]][j2,])^2)
}
}
print(length(which(d <= thres)))
d[which(d <= thres)] <- 1/d[which(d <= thres)]
prob <- exp(-d^2/2)
prob <- prob/sum(prob)
max.child <- pop.gen[i-1]*incr.ratio * 5
n.child <- sample(2:max.child,1,prob = dpois(2:max.child,pop.gen[i-1]*incr.ratio))
pop.gen <- c(pop.gen, n.child)
parents.number <- sample(1:length(d),pop.gen[i],replace=TRUE,prob = prob)
address <- matrix(1:length(d),pop.gen[i-1],pop.gen[i-1])
parents.list <- which(address <= max(address),arr.ind=TRUE)
parents[[i]] <- parents.list[parents.number,]
loc[[i]] <- (loc[[i-1]][parents[[i]][,1],] + loc[[i-1]][parents[[i]][,2],])/2
loc[[i]] <- loc[[i]] + cbind(sign(loc[[i]][,1])*rnorm(length(loc[[i]][,1]),rnorm(1,migration[1])),sign(loc[[i]][,2])*rnorm(length(loc[[i]][,2]),rnorm(1,migration[2])))
loc[[i]] <- loc[[i]] + cbind((loc[[i]][,2]),-(loc[[i]][,1])) /mean(abs(loc[[i]]))
}
xlim <- ylim <- range(unlist(loc))
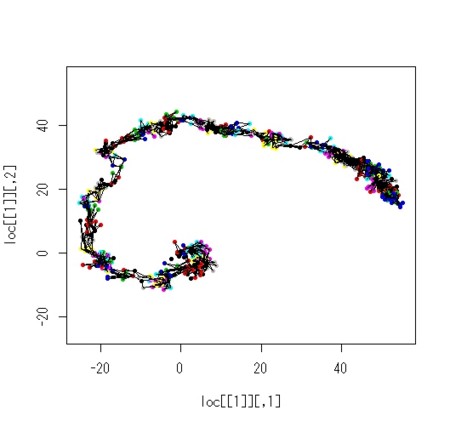
plot(loc[[1]],xlim = xlim, ylim = ylim,col=1,cex = 1,pch =20)
for(i in 2:n.gen){
points(loc[[i]],col=i,cex = 1,pch=20)
for(j in 1:pop.gen[i]){
segments(loc[[i]][j,1],loc[[i]][j,2],loc[[i-1]][parents[[i]][j,1],1],loc[[i-1]][parents[[i]][j,1],2])
segments(loc[[i]][j,1],loc[[i]][j,2],loc[[i-1]][parents[[i]][j,2],1],loc[[i-1]][parents[[i]][j,2],2])
Sys.sleep(0.01)
}
Sys.sleep(0.1)
}