- 昨日の続き
- 発現解析の結果をヒートマップ表示することがある
- Rではheatmap()関数がある
- heatmap()関数では、その内部で用いる距離定義関数(デフォルトはdist()関数)、階層的クラスタリング関数(デフォルトではhclust())を用いて、データ行列の行についてクラスタリング、列についてクラスタリング、出来た行クラスター、列クラスターを示すために行順序の入れ替え、列順序の入れ替えを実施し、その上で、行列のセルの値に色をつけて表示している
- heatmap()関数を使っての表示と、個別の処理をdist(),hclust()を使って実施して表示する手順とを併記してみる
- ヒートマップに適した行列データを作る
Ns <- 500
Nm <- 1000
Ns.pt <- 10
Nm.pt <- 10
trail <- matrix(rnorm(Ns.pt*Nm.pt),Ns.pt,Nm.pt)
trail <- apply(trail,2,cumsum)
library(rgl)
plot3d(trail[,1:3])
matplot(trail,type="l")
library(MCMCpack)
ps <- rdirichlet(1,rep(1,Ns.pt))
pm <- rdirichlet(1,rep(1,Nm.pt))
ss <- sample(1:Ns.pt,Ns,replace=TRUE,prob=ps)
sm <- sample(1:Nm.pt,Nm,replace=TRUE,prob=pm)
M <- trail[ss,sm]
M <- jitter(M,1000)

image(M)

heatmap(M)

d1 <-dist(M)
h1 <-hclust(d1)
plot(h1)

d2 <- dist(t(M))
h2 <- hclust(d2)
plot(h1)
clustered.M <- M[h1$order,h2$order]
-
- 配置換えした行列のセルに色を付けて表示
- クラスタリングは、枝分けを入れ替えてもクラスターの結果としては変わらないこともあるので、みてくれは完全に一致していないかもしれないが、情報提示的には同一とみなせる
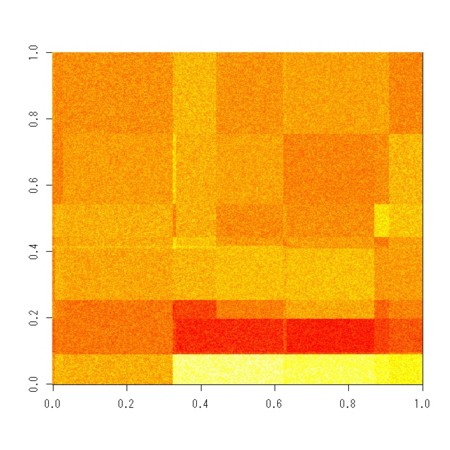
image(clustered.M)




